1.2. El modelo entidad-relación
Podemos estructurar cualquier realidad en diferentes entidades que pueden interactuar entre ellas siguiendo determinadas reglas. Por ejemplo, el genoma, en tanto que parte de la realidad biológica de una célula, también puede estructurarse en distintos componentes. A partir de esta organización artificialmente construida, podemos modelar la totalidad de los elementos que lo conforman utilizando entidades y relaciones. Estas estructuras, contenedores de información o datos pueden ser digitalizados en un ordenador para su gestión y análisis; en el caso del genoma, análisis bioinformáticos.
El modelo entidad-relación nos ayuda a diseñar nuestra propia base de datos. Una entidad representa una clase de elementos en el entorno real que deseamos modelar. Una ocurrencia es una instancia o ejemplo particular de una entidad. La conectividad o participación entre entidades debe especificarse explícitamente mediante relaciones. El número de ocurrencias de una entidad que podrán relacionarse con instancias de otra entidad será:
- Uno a uno (1:1): un elemento de la primera entidad puede relacionarse con un único elemento de la segunda.
- Uno a varios (1:N): un elemento de la primera entidad puede relacionarse con varios elementos de la segunda (pero no al contrario).
- Varios con varios (M:N): un elemento de la primera entidad puede relacionarse con varios elementos de la segunda (y viceversa).
Fuente: elaboración propia.
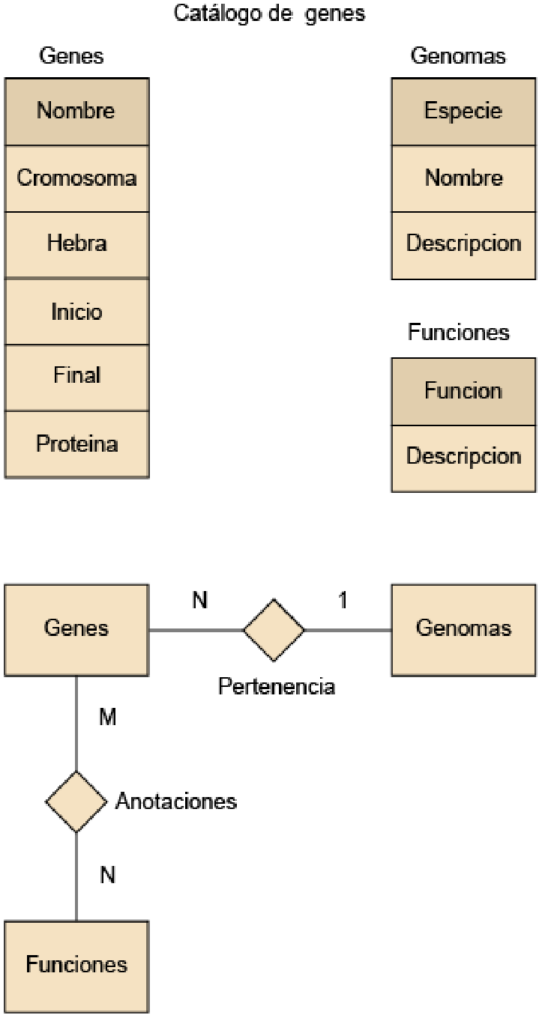
El modelo contiene tres entidades y dos relaciones. Gráficamente, las entidades se representan utilizando rectángulos, y las relaciones, mediante líneas rectas con un rombo para indicar la conectividad.
Este esquema basado en entidades y relaciones resulta sencillo de emplear posteriormente para construir la base de datos definitiva. Para mostrar las diferentes etapas de diseño, procederemos a modelar un escenario muy habitual en entornos de investigación bioinformáticos: la caracterización del catálogo de genes de un genoma. Podéis observar las diferentes entidades con sus atributos y las relaciones entre entidades que van a formar nuestro modelo en la figura 1.
Los genes son fragmentos de ADN ubicados en una localización precisa del genoma que codifican la secuencia de una proteína. Estas moléculas, por otro lado, desempeñan una función biológica específica dentro del organismo. Lógicamente, cada genoma posee su propio catálogo de genes. Analizando esta información previa, decidimos modelar este entorno utilizando las entidades genes, genomas y funciones, con sus propios atributos (mostrados en la figura 2).
Lógicamente, las entidades no son objetos aislados de su entorno. Debemos, por tanto, cumpliendo las especificaciones de nuestro problema, unir mediante relaciones aquellas entidades que están interconectadas en el mundo real. En este caso, los genes poseen la capacidad de pertenecer a un genoma para desarrollar una función concreta en el organismo. Para satisfacer ambas propiedades, definimos las relaciones binarias pertenencia y anotaciones, cada una con una conectividad diferente (figura 1). Las entidades y relaciones que forman este modelo deben ser convertidas en tablas. Las entidades y sus atributos pasarán a ser tablas de nuestra base de datos, pero no todas las relaciones van a ser tablas, va a depender del tipo de conectividad. Es básico establecer para cada tabla un atributo especial (o una combinación de ellos) que identifique cada instancia de forma unívoca. Este atributo especial recibe el nombre de clave primaria. Es preferible usar un código característico en lugar de un nombre para facilitar la identificación de cualquier instancia mediante la clave primaria (por ejemplo, un código numérico asignado en función del orden de entrada en la tabla).

Hemos subrayado la clave primaria de cada tabla.
Fuente: elaboración propia.
Las dos relaciones existentes en nuestro esquema deben modelarse de distinta forma, debido a que cada una presenta una combinación diferente de posibles ocurrencias entre tablas. La asociación pertenencia entre las tablas genes y genomas debe representarse con una relación con cardinalidad 1:N, pues un gen solo pertenece a un genoma, pero un genoma contiene muchos genes. Esta relación no necesita una tabla nueva, puede implementarse referenciando simplemente desde una tabla (genes), que es la parte n de la relación pertenencia, la clave primaria de la otra (genomas) que es la parte 1 de la relación pertenencia. Dentro de la tabla genes, este atributo recibe la denominación de clave foránea.

Indicamos con un subrayado superior la clave foránea de cada tabla.
Fuente: elaboración propia.
La relación anotaciones entre las tablas genes y funciones tiene una conectividad M:N, pues un gen puede poseer varias anotaciones, pero una anotación también puede ser compartida por varios genes. Para su correcta implementación, introduciremos una nueva tabla llamada anotaciones. Esta poseerá, como clave primaria la combinación de las claves primarias de cada tabla original.
Dado que ambas claves primarias por separado poseen todas las propiedades necesarias, el diseñador garantiza con esta medida que cada instancia de esta nueva tabla estará dotada de un identificador único, que estará formado por el nombre del gen junto con el nombre de la función en particular para que sea un identificador de la instancia único que no se pueda repetir.

Fuente: elaboración propia.
Las relaciones con cardinalidad M:N exportadas desde el modelo entidad-relación están caracterizadas también por sus propios atributos. Así, es posible añadir un campo para guardar el origen de cada anotación (por ejemplo, computacional, experimental o fuente bibliográfica). Podemos extraer esta información a partir de los datos recuperados del sistema de anotación automática utilizado para poblar de ejemplos nuestro catálogo de genes:

Fuente: elaboración propia.
La selección de las claves idóneas resulta esencial dentro del diseño de una base de datos relacional. De hecho, la integridad de un modelo relacional debe cumplir dos requisitos fundamentales relacionados con la gestión de estas:
- La clave primaria no debe contener un valor indefinido o nulo y el valor ha de ser único.
- La clave foránea debe hacer referencia a una clave primaria de otra tabla.